Ferramentas de análise das propriedades da proteína
De modo a se descobrir quais os genes que interessavam, ou seja, averiguar quais os que eram ou não funcionais, procedeu-se a uma recolha manual de propriedades, no NCBI e no uniProt. Para tal, no NCBI, limitou-se a pesquisa à zona de estudo pretendida e procedeu-se à pesquisa gene a gene (Versão GenBank). Devido à alteração no enunciado, procedeu-se à procura da informação pretendida com mais rigor.
Na tabela que se segue em anexo, estão referidos os seguintes asarâmetros, encontrados para caracterizar as proteínas codificads:
-
Gene Identification (Name, Locu Tag, NCBI GeneID, NCBI Acesion (GI), Strand)
-
Protein Identification (Name, NCBI Acession Number, Uniprot ID, Uniprot Acession Number, Revision)
-
Protein Properties (Amino Acids Number, Cellular Localization, Atomic Mass (Da), Conserved Domains)
-
Gene Ontology associated terms
-
EC Number
-
TC Number (embora na nossa zona genómica não exista)
-
Description (Funções das proteínas, nomeadamente por categorias)
-
Notes (Notas do NCBI)
www.dropbox.com/s/8ykdggeg37m2qpu/AnaliseProteinas.xlsx?dl=0
Após a elaboração desta tabela conclui-se que uma pesquisa mais profunda como a realizada permitiu adquirir mais conhecimentos à cerca das propriedades das proteínas. Constatou-se que as funções maioritariamente encontradas são: proteínas como Transportadoras e proteínas como Fatores de Regulação (destacando-se no processo de Transcrição).
NOTA: O ficheiro GenBank utilizado encontra-se no tópico "Análise de Sequência".
Script SwissProt
A SwissProt é uma base de dados composta apenas por registos curados, possuindo anotações de maior fiabilidade, visto que os seus conteúdos são cuidadosamente revistos. O objetivo deste script consiste em aceder à SwissProt e retirar algumas informações de interesse.
Deste modo, procedeu-se à verificação manual dos genes da zona do genoma de interesse que continham registo na Swissprot e guardou-se as identificações existentes num dicionário. Conhecendo as identificações , utilizou-se a função get_sprot_raw() para aceder à base de dados.
Criou-se um objeto SwissProt.Record para cada gene de interesse e exportaram-se algumas das informações relevantes para um ficheiro (my_swissprot.txt).
O código desenvolvido para a construção do script e o ficheiro obtido pela execução deste encontram-se abaixo.
Script "SwissPROT.py"
www.dropbox.com/s/sdpye8w2xcwsfnx/swissPROT.py?dl=0
Ficheiro obtido "my_swissprot.txt"
Alteração do mapeamento inicial
Inicialmente construiu-se um mapeamento de genes baseado apenas na anotação funcional presente nas features. Porém, depois de uma pesquisa mais aprofundada na base de dados UniProt conseguiram-se identificar as funções dos genes que, segundo a informação das features, não possuiam função. Note-se que caso se tivesse pesquisado em mais base de dados, provávelmente encontrariam-se outras funções e mais funções.
De um total de 97 genes inicialmente sem função definida, encontraram-se prováveis funções para 15 deles, ficando assim, de entre os 216 genes, 82 sem função conhecida e 134 com provável função. O mapeamento final dos genes e respetiva legenda encontram-se nas figuras seguintes. Note-se que foi realizada uma nova tabela, tendo em conta os tópicos fornecidos pelo docente para o enunciado complementar.
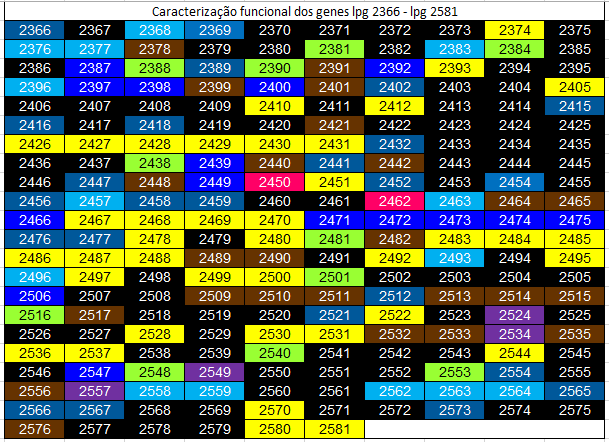
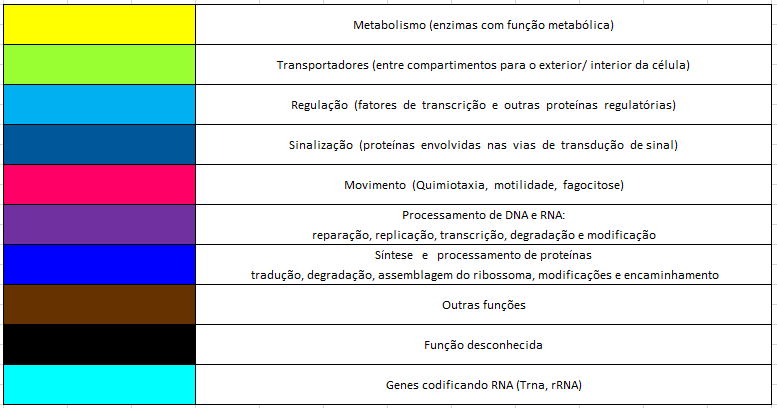
Catalogando os genes através da sua ação funcional, tem-se: 39 genes com função metabólica, 11 genes com função transportadora, 14 genes com função regulatória, 24 genes com função de sinalização, 2 genes associado ao movimento, 4 genes com funções em termos de processamento de DNA e RNA, 15 genes que entram na síntese e processamento de proteínas, 25 genes com outras funções e por fim, 82 genes sem função conhecida, dando isto um total de 216 genes.
